研究概要
MALDI-TOF MSを用いた新規な微生物分類法-S10-GERMS法
遺伝子による分類が困難な微生物の迅速・簡便同定法の開発
微生物を分類・同定する手法は,これまでに様々な方法が報告されており,また新しい方法も考案されています.しかし,それぞれの方法には一長一短があり,また適用できる範囲が限られることがあります.そこで,現在の分類学では多面的な方向からのデータを総合的に判断する多相分類学の概念が取り入れられています.すなわち,分類学的な検討を行う場合には,類縁菌種との鑑別に有用な種々の手法を実施し,総合的に判断します.細菌を分類・同定するための方法としては大きく分けて,以下の5つに分けられます.
1.形態観察
2.表現形質(生理・生化学性状)
3.化学分類指標(細胞壁組成, キノン分子種など)
4.蛋白分析(全菌体蛋白組成など)
5.DNA 分析(G+C mol%, DNA 相同試験, 塩基配列比較など)
これら各分類方法は換言すると,表現型(phenotype)による方法と遺伝子型(genotype)による方法に分けることができます.
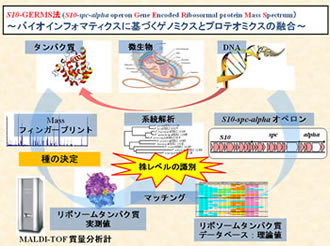
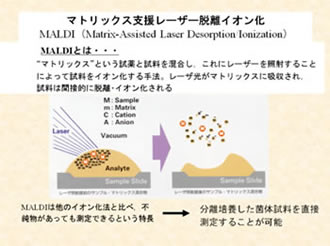
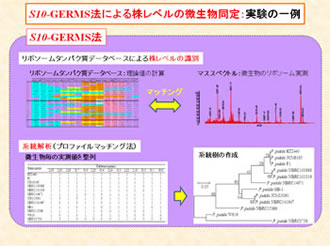
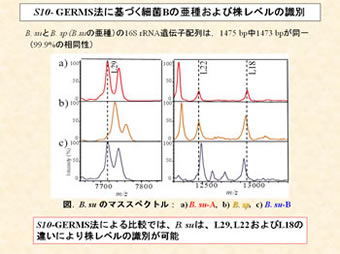
現在のDNA分析では,16S rRNA遺伝子配列のデータが蓄積され系統解析が容易にできるようになったため,16S rRNA遺伝子配列による微生物同定が簡易同定法としての主流となっています. 16S rRNA遺伝子の塩基配列データの適用範囲は,科以上のレベルから属・種の推定と広範囲なため,供試菌体がどの属あるいは種と類縁関係にあるか推測できます.16S rRNA遺伝子は,全生物(真核生物では18S rRNA)に共通に存在し,その進化速度が比較的遅く,系統の離れた生物間での比較も容易にでき,系統学的位置を多様な細菌の中から推定するには適当です.一方で進化速度が遅いため,非常に近縁な菌種間での比較は困難となります.このような場合には,進化速度の速い遺伝子(gyrB, rpoD, dnaJなど)の塩基配列を決定し,より詳細な系統解析を行う方法もあります.環境微生物や有用微生物の選抜では,分類学的な試験項目を簡略化して数多くの菌株を分類・同定する必要があります.また,食中毒が起こった場合等では原因となる病原菌の特定には一刻を争う場合もあります.このように通常の手法では,細菌を取り扱う現場での分析には対応しきれない場合が多々あることから,迅速・簡便かつ信頼性の高い細菌の同定手法の開発が求められています.そこで本研究室では,新しい微生物同定法を開発するために細菌の全ゲノムデータベースを用いたバイオインフォマティクスに基づくバイオマーカータンパク質のMALDI-TOF MS(マトリックス支援レーザー脱離イオン化-時間飛行型 質量分析計)による微生物の同定(S10–spc–alpha operon Gene Encoded Ribosomal protein Mass Spectrum : S10–GERMS法)に関する研究を行っています.これにより,従来の方法では達成できなかった株レベルでの細菌種の識別ができると考えています.この研究成果は,学会での発表でポスター賞を受賞し,招待講演などにより評価を受けています.それにより細菌の簡便・迅速な同定・識別システムとして,(株)島津製作所を通じて試験的に市場展開しています.
|
|

|
|
|
 |
|
本研究成果は,2013/9/3から(株)島津製作所より「AXIMA微生物同定システム対応高精度細菌識別ソフトウェア Strain Solution」として販売が開始されました。 |
|


